Segmentação celular: o que é e como a Visão de IA aprimora
Aprenda como funciona a segmentação de células e como a Visão de IA melhora a análise de microscopia com aprendizado profundo, métricas-chave, conjuntos de dados e usos no mundo real.
Aprenda como funciona a segmentação de células e como a Visão de IA melhora a análise de microscopia com aprendizado profundo, métricas-chave, conjuntos de dados e usos no mundo real.

Muitos avanços na descoberta de medicamentos, na investigação do cancro ou na medicina personalizada começam com um desafio fundamental: ver as células com clareza. Os cientistas dependem de imagens nítidas para track comportamento das células, avaliar medicamentos e explorar novas terapias.
Uma única imagem de microscopia pode conter milhares de células sobrepostas, dificultando a visualização dos limites. A segmentação de células visa resolver isso, separando claramente cada célula para uma análise precisa.
Mas a segmentação de células nem sempre é simples. Um único estudo pode produzir milhares de imagens detalhadas de microscópio, muitas para serem revisadas manualmente. À medida que os conjuntos de dados crescem, os cientistas precisam de maneiras mais rápidas e confiáveis de separar e estudar as células.
De facto, muitos cientistas estão a adotar a visão computacional, um ramo da IA que permite às máquinas interpretar e analisar informações visuais. Por exemplo, modelos como o Ultralytics YOLO11 que suportam a segmentação de instâncias, podem ser treinados para separar células e até detect estruturas subcelulares. Isto permite uma análise precisa em segundos, em vez de horas, ajudando os investigadores a escalar os seus estudos de forma eficiente.
Neste artigo, exploraremos como funciona a segmentação de células, como a visão computacional a aprimora e onde ela é aplicada no mundo real. Vamos começar!
Tradicionalmente, os cientistas segmentavam as células à mão, traçando-as em imagens de microscopia. Isso funcionava bem para pequenos projetos, mas era lento, inconsistente e propenso a erros. Com milhares de células sobrepostas em uma única imagem, o rastreamento manual rapidamente se torna opressor e um grande gargalo.
A visão computacional oferece uma opção mais rápida e confiável. É um ramo da IA alimentado por aprendizado profundo, onde as máquinas aprendem padrões a partir de grandes conjuntos de imagens. Na pesquisa celular, isso significa que elas podem reconhecer e separar células individuais com alta precisão.
Especificamente, os modelos de IA de visão, como o Ultralytics YOLO11 , suportam tarefas como a deteção de objectos e a segmentação de instâncias, e podem ser treinados em conjuntos de dados personalizados para analisar células. A deteção de objectos permite encontrar e rotular cada célula de uma imagem, mesmo quando muitas aparecem juntas.
A segmentação de instâncias vai um passo além, desenhando limites precisos ao redor de cada célula, capturando suas formas exatas. A integração dessas capacidades de Visão de IA em pipelines de segmentação de células permite que os pesquisadores automatizem fluxos de trabalho complexos e processem imagens de microscopia de alta resolução de forma eficiente.
Os métodos de segmentação celular mudaram muito ao longo dos anos. As primeiras técnicas de segmentação de imagem funcionavam para imagens simples, mas apresentavam dificuldades à medida que os conjuntos de dados aumentavam e as células se tornavam mais difíceis de distinguir.
Para superar essas limitações, abordagens mais avançadas foram desenvolvidas, levando aos modelos de visão computacional atuais que trazem velocidade, precisão e escalabilidade para estudos de microbiologia e microscopia.
Em seguida, vamos percorrer a evolução dos algoritmos de segmentação, desde os métodos básicos de thresholding até os modelos de deep learning de ponta e os pipelines híbridos.
Antes dos avanços na visão computacional, a segmentação de células dependia de técnicas tradicionais de processamento de imagem. Esses métodos se baseavam em regras e operações definidas manualmente, como detecção de bordas, separação do primeiro plano do fundo ou suavização de formas. Ao contrário dos modelos de visão computacional, que podem aprender padrões diretamente dos dados, o processamento de imagem depende de algoritmos fixos aplicados da mesma forma em todas as imagens.
Uma das primeiras abordagens foi o thresholding, um método que separa as células do fundo comparando os níveis de brilho dos pixels. Isso funciona bem quando há um forte contraste entre as células e o seu entorno.
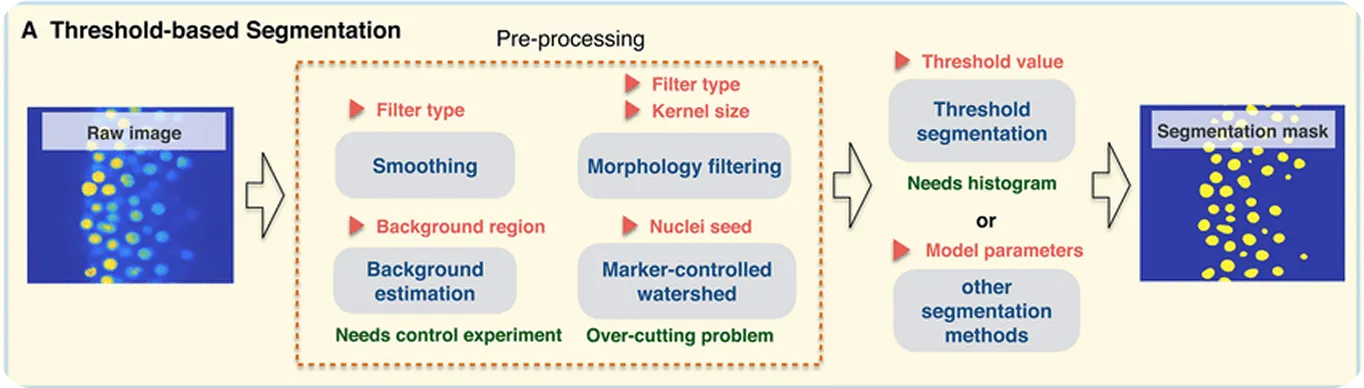
Para refinar os resultados, operações morfológicas como a dilatação (expansão de formas) e a erosão (contração de formas) são usadas para suavizar arestas, remover ruídos ou fechar pequenas lacunas. Para células que se tocam ou se sobrepõem, uma técnica chamada segmentação watershed ajuda a separá-las, traçando limites onde as células se encontram.
Embora essas técnicas tenham dificuldades com casos complexos, como células sobrepostas ou imagens ruidosas, elas ainda são úteis para aplicações mais simples e são uma parte importante da história da segmentação de células. Suas limitações, no entanto, impulsionaram o campo em direção a modelos baseados em deep learning, que oferecem uma precisão muito maior para imagens mais desafiadoras.
À medida que as técnicas de processamento de imagem atingiram os seus limites, a segmentação de células passou a adotar abordagens baseadas em aprendizagem. Ao contrário dos métodos baseados em regras, os modelos de aprendizagem profunda identificam padrões diretamente a partir dos dados, tornando-os mais adaptáveis a células sobrepostas, formas variáveis e diferentes modalidades de imagem.
As redes neuronais convolucionais (CNN) são uma classe de arquitecturas de aprendizagem profunda amplamente utilizadas na visão computacional. Processam imagens por camadas: as primeiras camadas detect caraterísticas simples, como arestas e texturas, enquanto as camadas mais profundas captam formas e estruturas mais complexas. Esta abordagem por camadas torna as CNNs eficazes para muitas tarefas visuais, desde o reconhecimento de objectos do quotidiano através do reconhecimento de padrões até à análise de imagens biomédicas.
Modelos como o YOLO11 são construídos com base nestes princípios de aprendizagem profunda. Alargam as arquitecturas baseadas em CNN com técnicas de deteção de objectos em tempo real e segmentação de instâncias, tornando possível localizar rapidamente as células e delinear os seus limites.
Pipelines híbridos melhoram a segmentação celular combinando os pontos fortes de vários métodos. Estes podem incluir processamento de imagem clássico e modelos de deep learning, ou mesmo diferentes modelos baseados em deep learning trabalhando juntos.
Por exemplo, um método pode melhorar ou pré-processar imagens de microscopia para reduzir o ruído e tornar os limites mais nítidos, enquanto outro modelo é utilizado para detect e segment as células. Ao dividir as tarefas desta forma, as abordagens híbridas melhoram a precisão, tratam imagens complexas de forma mais eficaz e tornam os estudos em grande escala mais fiáveis.

Outro fator chave a ser considerado na segmentação de células orientada por visão computacional são os dados de imagem. Os modelos de visão computacional dependem fortemente de conjuntos de dados grandes e de alta qualidade para aprender como identificar e separar células com precisão.
Esses conjuntos de dados normalmente consistem em imagens de microscopia emparelhadas com anotações. Tão importante quanto as imagens brutas, a anotação de dados, ou rotulagem, desempenha um papel crucial na criação de conjuntos de dados eficazes, pois informa ao modelo informações importantes.
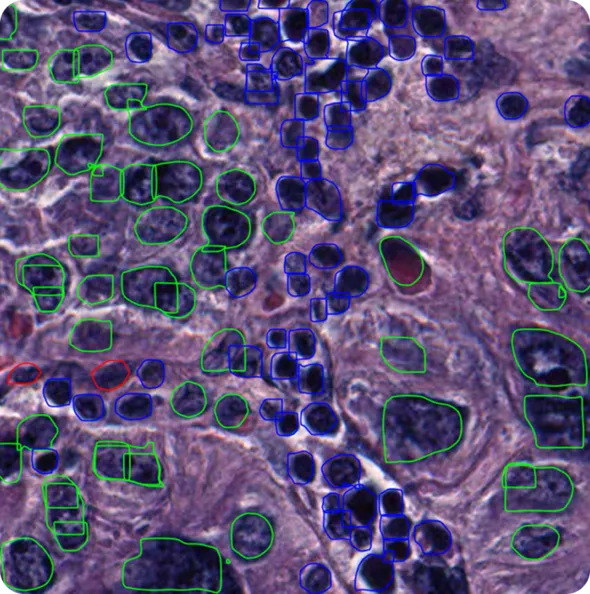
Por exemplo, se quisermos treinar um modelo como o YOLO11 para segment células cancerosas a partir de imagens de microscopia, precisamos de exemplos rotulados que mostrem onde cada célula começa e termina. Estas etiquetas funcionam como um guia, ensinando o modelo a reconhecer a morfologia e os limites das células. As anotações podem ser desenhadas à mão ou criadas com ferramentas semi-automatizadas para poupar tempo.
O tipo de anotação também depende da tarefa. Para detecção de objetos, caixas delimitadoras são desenhadas ao redor de cada célula. Para segmentação de instâncias, os rótulos são mais como máscaras detalhadas que traçam o contorno exato de cada célula. Escolher o tipo certo de anotação e dados de treinamento ajuda o modelo a aprender o que ele precisa para o trabalho.
Em geral, construir conjuntos de dados de visão computacional pode ser difícil, especialmente quando não existem coleções de imagens existentes ou quando o campo é muito único e especializado. Mas no campo da investigação celular, existem dificuldades técnicas que tornam a recolha e a anotação de dados ainda mais complexas.
As imagens de microscopia podem parecer muito diferentes dependendo do método de imagem celular. Por exemplo, a microscopia de fluorescência usa corantes que fazem com que partes de uma célula brilhem. Essas imagens de fluorescência destacam detalhes que, de outra forma, seriam difíceis de ver.
A anotação é outro grande desafio. Rotular milhares de células manualmente é lento e requer conhecimento especializado. As células frequentemente se sobrepõem, mudam de forma ou parecem fracas, facilitando a ocorrência de erros. Ferramentas semiautomáticas podem acelerar o processo, mas a supervisão humana geralmente é necessária para garantir a qualidade.
Para facilitar a carga de trabalho, os pesquisadores às vezes usam anotações mais simples, como marcadores de localização de interesse que indicam onde as células estão, em vez de desenhar contornos completos. Embora menos precisos, esses marcadores ainda fornecem orientação crucial para o treinamento.
Além disso, o compartilhamento de dados em biologia adiciona mais complicações. Preocupações com a privacidade, o consentimento do paciente e as diferenças nos equipamentos de imagem entre os laboratórios podem dificultar a construção de conjuntos de dados consistentes e de alta qualidade.
Apesar destes obstáculos, os conjuntos de dados de código aberto fizeram uma grande diferença. As coleções públicas partilhadas através de plataformas como o GitHub fornecem milhares de imagens rotuladas em muitos tipos de células e métodos de imagem, ajudando os modelos a generalizar melhor para cenários do mundo real.
Agora que temos uma melhor compreensão dos dados e métodos usados para segmentar células com visão computacional, vamos examinar algumas das aplicações no mundo real da segmentação de células e da visão computacional.
A análise de célula única ou o estudo de células individuais em vez de amostras de tecido inteiras ajuda os cientistas a ver detalhes que geralmente são perdidos em um nível mais amplo. Essa abordagem é amplamente utilizada em biologia celular, descoberta de medicamentos e diagnósticos para entender como as células funcionam e respondem sob diferentes condições.
Por exemplo, na pesquisa do câncer, uma amostra de tecido geralmente contém uma mistura de células cancerosas, células imunológicas e células de suporte (estromais). Olhar apenas para o tecido como um todo pode esconder diferenças importantes, como a forma como as células imunológicas interagem com os tumores ou como as células cancerosas perto dos vasos sanguíneos se comportam.
A análise de uma única célula permite aos investigadores separar estes tipos de células e estudá-las individualmente, o que é crucial para compreender as respostas ao tratamento e a progressão da doença. Modelos como o YOLO11, que suportam a segmentação de instâncias, podem detect cada célula e delinear a sua forma exacta, mesmo em imagens sobrepostas ou com muita gente. Ao transformar imagens de microscopia complexas em dados estruturados, YOLO11 permite aos investigadores analisar milhares de células de forma rápida e consistente.
As células se dividem, movem e respondem ao ambiente de maneiras diferentes. Analisar como as células vivas mudam ao longo do tempo ajuda os cientistas a entender como elas se comportam em condições de saúde e doença.
Com ferramentas como o contraste de fase ou a microscopia de alta resolução, os investigadores podem acompanhar estas alterações sem adicionar corantes ou etiquetas. Isto mantém as células no seu estado natural e torna os resultados mais fiáveis.
Rastrear células ao longo do tempo também ajuda a capturar detalhes que poderiam ser perdidos. Uma célula pode mover-se mais rápido do que outras, dividir-se de uma forma incomum ou responder fortemente a um estímulo. Registar estas diferenças subtis fornece uma imagem mais clara de como as células se comportam em condições reais.
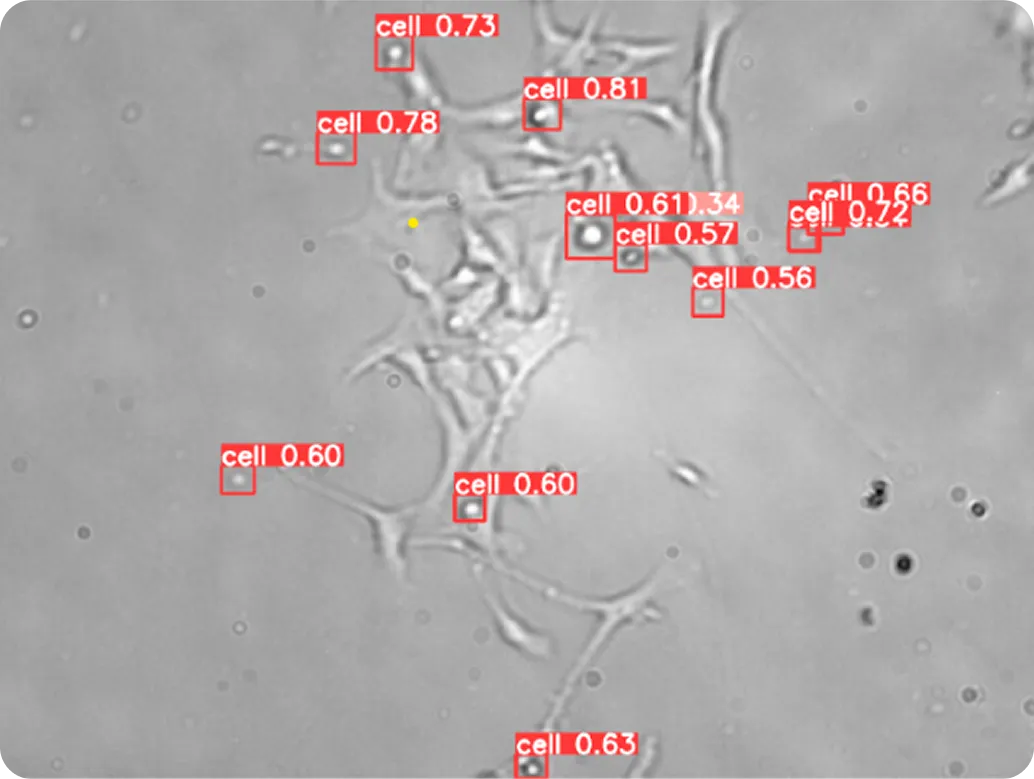
Modelos de visão por computador, tais como Ultralytics YOLOv8 tornam este processo mais rápido e mais consistente. Ao detetar e seguir células individuais através de sequências de imagens, YOLOv8 pode monitorizar automaticamente os movimentos, divisões e interações celulares, mesmo quando as células se sobrepõem ou mudam de forma.
Os conhecimentos obtidos a partir de modelos de visão por computador, como o YOLO11 , utilizados para a segmentação de células inteiras, podem ter um maior impacto quando combinados com a bioinformática (a utilização de métodos computacionais para analisar dados biológicos) e a multiómica (a integração de informações sobre ADN, ARN e proteínas). Em conjunto, estes métodos fazem com que a investigação vá para além da definição dos limites das células e passe a compreender o significado desses limites.
Em vez de apenas identificar onde as células estão, os cientistas podem estudar como elas interagem, como a estrutura do tecido muda na doença e como pequenas mudanças na forma da célula se conectam à atividade molecular.
Considere uma amostra de tumor: ao associar o tamanho, a forma ou a posição das células cancerígenas aos seus perfis moleculares, os investigadores podem encontrar correlações com mutações genéticas, expressão genética ou atividade proteica anormal. Isto transforma imagens estáticas em conhecimentos práticos, ajudando a track atividade dos genes nos tumores, a mapear o comportamento das proteínas em tempo real e a construir atlas de referência que ligam a estrutura à função.
Aqui estão alguns dos principais benefícios de usar a visão computacional para a segmentação de células:
Embora a visão computacional traga muitos benefícios para a segmentação de células, ela também tem algumas limitações. Aqui estão alguns fatores a serem considerados:
A próxima geração de segmentação de células provavelmente será definida por modelos de visão computacional que combinam velocidade, precisão e escalabilidade. Modelos como o U-Net têm sido altamente influentes, mas podem ser computacionalmente exigentes. Com os avanços na visão computacional, os pesquisadores estão migrando para modelos que oferecem alta precisão e desempenho em tempo real.
Por exemplo, os modelos topo de gama, como o Ultralytics YOLOv8 , podem segment imagens de microscopia muito mais rapidamente do que as abordagens tradicionais, produzindo simultaneamente limites nítidos e precisos.
Num estudo recente de Microscopia Eletrónica de Transmissão (TEM), as métricas de desempenho mostraram que YOLOv8 funcionou até 43 vezes mais rápido do que a U-Net. Este tipo de desempenho torna possível analisar grandes conjuntos de dados em tempo real, o que é cada vez mais importante à medida que os estudos de imagiologia aumentam de tamanho.

Estas melhorias já estão a ser postas em prática. Plataformas como o Theiascope™ da Theia Scientific integram os modelos Ultralytics YOLO com a Microscopia Eletrónica de Transmissão (TEM), permitindo que as estruturas em nanoescala sejam segmentadas de forma consistente e em escala. A plataforma utiliza modelos Ultralytics YOLO para deteção e segmentação em tempo real, identificando automaticamente estruturas em imagens TEM à medida que são capturadas e convertendo-as em dados fiáveis e prontos a analisar.
A segmentação celular desempenha um papel fundamental na microscopia moderna e na investigação biomédica. Permite aos cientistas observar células individuais, track a progressão de doenças e monitorizar a forma como os tratamentos afectam o comportamento das células. Os modelos de IA de visão como o YOLO11 tornam este processo mais rápido e mais preciso. Ao lidar facilmente com imagens grandes e complexas, garantem que as experiências são repetíveis e escaláveis.
Junte-se à nossa comunidade e visite nosso repositório GitHub para saber mais sobre IA. Explore nossas páginas de soluções para saber mais sobre aplicações como IA na agricultura e visão computacional na logística. Confira nossas opções de licenciamento e comece a construir com visão computacional hoje mesmo!