Zellsegmentierung: Was sie ist und wie Vision AI sie verbessert
Erfahren Sie, wie Zellsegmentierung funktioniert und wie Vision AI die Mikroskopieanalyse mit Deep Learning, Schlüsselmetriken, Datensätzen und realen Anwendungen verbessert.
Erfahren Sie, wie Zellsegmentierung funktioniert und wie Vision AI die Mikroskopieanalyse mit Deep Learning, Schlüsselmetriken, Datensätzen und realen Anwendungen verbessert.

Viele Durchbrüche in der Arzneimittelforschung, der Krebsforschung oder der personalisierten Medizin beginnen mit einer zentralen Herausforderung: Zellen klar zu sehen. Wissenschaftler sind auf klare Bilder angewiesen, um das Zellverhalten track , Medikamente zu bewerten und neue Therapien zu erforschen.
Ein einzelnes Mikroskopiebild kann Tausende von sich überlappenden Zellen enthalten, wodurch die Grenzen schwer zu erkennen sind. Die Zellsegmentierung zielt darauf ab, dies zu lösen, indem jede Zelle für eine genaue Analyse klar getrennt wird.
Zellsegmentierung ist jedoch nicht immer einfach. Eine einzelne Studie kann Tausende von detaillierten Mikroskopiebildern erzeugen, viel zu viele, um sie manuell zu überprüfen. Mit wachsenden Datensätzen benötigen Wissenschaftler schnellere und zuverlässigere Methoden, um Zellen zu trennen und zu untersuchen.
Viele Wissenschaftler setzen auf Computer Vision, einen Zweig der KI, der es Maschinen ermöglicht, visuelle Informationen zu interpretieren und zu analysieren. Zum Beispiel sind Modelle wie Ultralytics YOLO11 die die Segmentierung von Instanzen unterstützen, können trainiert werden, um Zellen zu trennen und sogar subzelluläre Strukturen detect . Dies ermöglicht eine präzise Analyse in Sekunden statt in Stunden und hilft den Forschern, ihre Studien effizient zu skalieren.
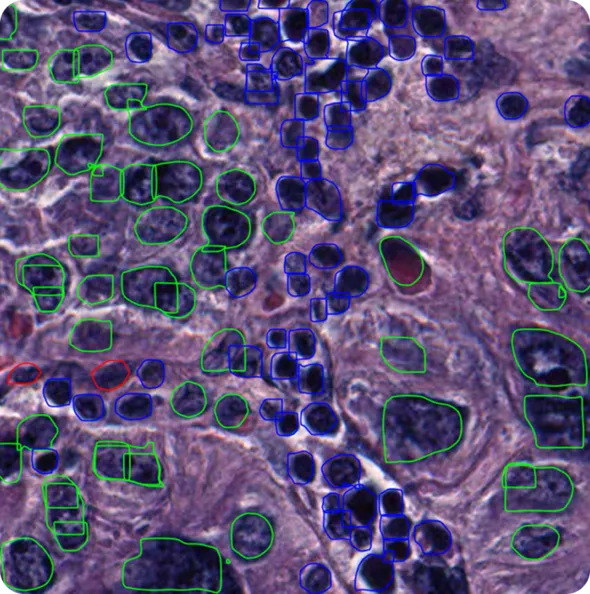
In diesem Artikel werden wir untersuchen, wie Zellsegmentierung funktioniert, wie Computer Vision sie verbessert und wo sie in der realen Welt eingesetzt wird. Los geht's!
Traditionell segmentierten Wissenschaftler Zellen von Hand, indem sie diese in Mikroskopiebildern nachzeichneten. Dies funktionierte gut für kleine Projekte, war aber langsam, inkonsistent und fehleranfällig. Bei Tausenden von überlappenden Zellen in einem einzigen Bild wird die manuelle Nachverfolgung schnell überwältigend und zu einem großen Engpass.
Computer Vision bietet eine schnellere und zuverlässigere Option. Es ist ein Zweig der KI, der auf Deep Learning basiert, bei dem Maschinen Muster aus großen Bildersätzen lernen. In der Zellforschung bedeutet dies, dass sie einzelne Zellen mit hoher Genauigkeit erkennen und trennen können.
Konkret unterstützen KI-Modelle wie Ultralytics YOLO11 Aufgaben wie Objekterkennung und Instanzsegmentierung und können auf benutzerdefinierten Datensätzen zur Analyse von Zellen trainiert werden. Die Objekterkennung ermöglicht es, jede einzelne Zelle in einem Bild zu finden und zu beschriften, auch wenn viele zusammen auftreten.
Die Instanzsegmentierung geht noch einen Schritt weiter, indem sie präzise Grenzen um jede Zelle zieht und deren exakte Form erfasst. Die Integration dieser Vision AI-Funktionen in Zellsegmentierungs-Pipelines ermöglicht es Forschern, komplexe Arbeitsabläufe zu automatisieren und hochauflösende Mikroskopiebilder effizient zu verarbeiten.
Die Methoden der zellulären Segmentierung haben sich im Laufe der Jahre stark verändert. Frühe Bildsegmentierungstechniken funktionierten bei einfachen Bildern, hatten aber Schwierigkeiten, als die Datensätze größer wurden und Zellen schwerer zu unterscheiden waren.
Um diese Einschränkungen zu überwinden, wurden fortschrittlichere Ansätze entwickelt, die zu den heutigen Computer-Vision-Modellen führten, welche Geschwindigkeit, Genauigkeit und Skalierbarkeit in mikrobiologische und mikroskopische Studien einbringen.
Als Nächstes wollen wir die Entwicklung der Segmentierungsalgorithmen durchgehen, von einfachen Schwellenwertmethoden bis hin zu hochmodernen Deep-Learning-Modellen und hybriden Pipelines.
Vor den Fortschritten in der Computer Vision basierte die Zellsegmentierung auf traditionellen Bildverarbeitungstechniken. Diese Methoden basierten auf manuell definierten Regeln und Operationen, wie z. B. dem Erkennen von Kanten, dem Trennen des Vordergrunds vom Hintergrund oder dem Glätten von Formen. Im Gegensatz zu Computer-Vision-Modellen, die Muster direkt aus Daten lernen können, hängt die Bildverarbeitung von festen Algorithmen ab, die auf alle Bilder auf die gleiche Weise angewendet werden.
Einer der frühesten Ansätze war Thresholding, eine Methode, die Zellen vom Hintergrund trennt, indem sie die Helligkeitswerte der Pixel vergleicht. Dies funktioniert gut, wenn ein starker Kontrast zwischen Zellen und ihrer Umgebung besteht.
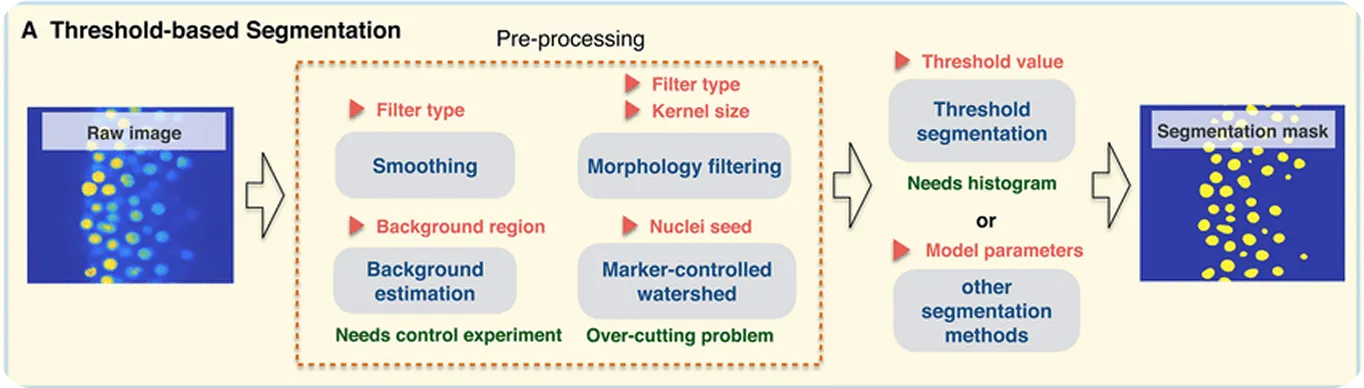
Um die Ergebnisse zu verfeinern, werden morphologische Operationen wie Dilatation (Erweitern von Formen) und Erosion (Verkleinern von Formen) verwendet, um Kanten zu glätten, Rauschen zu entfernen oder kleine Lücken zu schließen. Für Zellen, die sich berühren oder überlappen, hilft eine Technik namens Watershed-Segmentation, diese zu trennen, indem Grenzen dort gezogen werden, wo sich die Zellen treffen.
Obwohl diese Techniken bei komplexen Fällen wie überlappenden Zellen oder verrauschten Bildern Schwierigkeiten haben, sind sie dennoch für einfachere Anwendungen nützlich und ein wichtiger Teil der Geschichte der Zellsegmentierung. Ihre Einschränkungen trieben das Feld jedoch in Richtung Deep Learning-basierter Modelle, die eine viel höhere Genauigkeit für anspruchsvollere Bilder liefern.
Als die Bildverarbeitungstechniken an ihre Grenzen stießen, verlagerte sich die Zellsegmentierung auf lernbasierte Ansätze. Im Gegensatz zu regelbasierten Methoden identifizieren Deep-Learning-Modelle Muster direkt aus Daten, wodurch sie besser an überlappende Zellen, variable Formen und verschiedene Bildgebungsmodalitäten angepasst werden können.
Faltungsneuronale Netze (Convolutional Neural Networks, CNNs) sind eine Klasse von Deep-Learning-Architekturen, die in der Computer Vision weit verbreitet sind. Sie verarbeiten Bilder in Schichten: Die ersten Schichten detect einfache Merkmale wie Kanten und Texturen, während tiefere Schichten komplexere Formen und Strukturen erfassen. Durch diesen schichtweisen Ansatz sind CNNs für viele visuelle Aufgaben geeignet, von der Erkennung von Alltagsgegenständen über die Mustererkennung bis hin zur Analyse biomedizinischer Bilder.
Modelle wie YOLO11 basieren auf diesen Prinzipien des Deep Learning. Sie erweitern CNN-basierte Architekturen um Techniken zur Objekterkennung und Instanzsegmentierung in Echtzeit, die es ermöglichen, Zellen schnell zu lokalisieren und ihre Grenzen zu umreißen.
Hybride Pipelines verbessern die Zellsegmentierung, indem sie die Stärken verschiedener Methoden kombinieren. Dazu können klassische Bildverarbeitung und Deep-Learning-Modelle gehören, oder sogar verschiedene Deep-Learning-basierte Modelle, die zusammenarbeiten.
So kann eine Methode beispielsweise Mikroskopiebilder verbessern oder vorverarbeiten, um Rauschen zu reduzieren und Grenzen zu schärfen, während ein anderes Modell zur detect und segment der Zellen verwendet wird. Durch diese Aufgabenteilung verbessern hybride Ansätze die Genauigkeit, verarbeiten komplexe Bilder effektiver und machen groß angelegte Studien zuverlässiger.

Ein weiterer wichtiger Faktor, der bei der computergestützten Zellsegmentierung zu berücksichtigen ist, sind die Bilddaten. Computer-Vision-Modelle sind in hohem Maße auf große, qualitativ hochwertige Datensätze angewiesen, um zu lernen, wie man Zellen genau identifiziert und trennt.
Diese Datensätze bestehen typischerweise aus Mikroskopiebildern, die mit Annotationen versehen sind. Genauso wichtig wie die Rohbilder ist die Datenannotation oder -kennzeichnung, die eine entscheidende Rolle bei der Erstellung effektiver Datensätze spielt, da sie dem Modell wichtige Informationen liefert.
Wenn wir zum Beispiel ein Modell wie YOLO11 trainieren wollen, um Krebszellen aus Mikroskopbildern segment , brauchen wir beschriftete Beispiele, die zeigen, wo jede Zelle beginnt und endet. Diese Beschriftungen dienen als Leitfaden, der dem Modell beibringt, wie es die Zellmorphologie und -grenzen erkennen kann. Die Beschriftungen können von Hand gezeichnet oder mit halbautomatischen Tools erstellt werden, um Zeit zu sparen.
Die Art der Annotation hängt auch von der Aufgabe ab. Für die Objekterkennung werden Begrenzungsrahmen um jede Zelle gezeichnet. Für die Instanzsegmentierung sind die Labels eher detaillierte Masken, die den genauen Umriss jeder Zelle nachzeichnen. Die Wahl der richtigen Art von Annotation und Trainingsdaten hilft dem Modell zu lernen, was es für die Aufgabe benötigt.
Im Allgemeinen kann das Erstellen von Computer-Vision-Datensätzen schwierig sein, insbesondere wenn keine vorhandenen Bildersammlungen vorhanden sind oder wenn das Feld sehr einzigartig und spezialisiert ist. Aber im Bereich der Zellforschung gibt es technische Schwierigkeiten, die die Datenerfassung und -annotation noch komplexer machen.
Mikroskopiebilder können je nach Zellbildgebungsverfahren sehr unterschiedlich aussehen. Beispielsweise verwendet die Fluoreszenzmikroskopie Farbstoffe, die Teile einer Zelle zum Leuchten bringen. Diese Fluoreszenzbilder heben Details hervor, die sonst schwer zu erkennen sind.
Die Annotation ist eine weitere große Herausforderung. Das manuelle Beschriften von Tausenden von Zellen ist langsam und erfordert Fachwissen. Zellen überlappen sich oft, ändern ihre Form oder erscheinen schwach, wodurch sich leicht Fehler einschleichen können. Halbautomatische Werkzeuge können den Prozess beschleunigen, aber eine menschliche Aufsicht ist in der Regel erforderlich, um die Qualität zu gewährleisten.
Um die Arbeitslast zu verringern, verwenden Forscher manchmal einfachere Annotationen, wie z. B. Ortsmarkierungen von Interesse, die angeben, wo sich Zellen befinden, anstatt vollständige Umrisse zu zeichnen. Obwohl diese Markierungen weniger präzise sind, bieten sie dennoch wichtige Anleitungen für das Training.
Darüber hinaus erschwert der Datenaustausch in der Biologie die Situation zusätzlich. Bedenken hinsichtlich des Datenschutzes, die Einwilligung der Patienten und Unterschiede in der Bildgebungsausrüstung zwischen den Laboren können es erschweren, konsistente, qualitativ hochwertige Datensätze zu erstellen.
Trotz dieser Hindernisse haben Open-Source-Datensätze einen großen Unterschied gemacht. Öffentliche Sammlungen, die über Plattformen wie GitHub geteilt werden, bieten Tausende von beschrifteten Bildern aus vielen Zelltypen und Bildgebungsverfahren und helfen Modellen, sich besser an reale Szenarien anzupassen.
Nachdem wir nun ein besseres Verständnis der Daten und Methoden zur Segmentierung von Zellen mit Computer Vision haben, wollen wir uns einige der realen Anwendungen der Zellsegmentierung und Computer Vision ansehen.
Die Einzelzellanalyse, bei der einzelne Zellen anstelle von ganzen Gewebeproben untersucht werden, hilft Wissenschaftlern, Details zu erkennen, die auf breiterer Ebene oft übersehen werden. Dieser Ansatz wird häufig in der Zellbiologie, der Wirkstoffforschung und der Diagnostik eingesetzt, um zu verstehen, wie Zellen unter verschiedenen Bedingungen funktionieren und reagieren.
Beispielsweise enthält eine Gewebeprobe in der Krebsforschung oft eine Mischung aus Krebszellen, Immunzellen und unterstützenden (stromalen) Zellen. Wenn man nur das Gewebe als Ganzes betrachtet, können wichtige Unterschiede verborgen bleiben, z. B. wie Immunzellen mit Tumoren interagieren oder wie sich Krebszellen in der Nähe von Blutgefäßen verhalten.
Die Einzelzellanalyse ermöglicht es den Forschern, diese Zelltypen zu trennen und einzeln zu untersuchen, was für das Verständnis der Behandlungsreaktionen und des Krankheitsverlaufs von entscheidender Bedeutung ist. Modelle wie YOLO11, die die Segmentierung von Instanzen unterstützen, können jede Zelle detect und ihre genaue Form umreißen, selbst in überfüllten oder überlappenden Bildern. Indem YOLO11 komplexe Mikroskopiebilder in strukturierte Daten umwandelt, ermöglicht es Forschern, Tausende von Zellen schnell und konsistent zu analysieren.
Zellen teilen sich, bewegen sich und reagieren auf unterschiedliche Weise auf ihre Umgebung. Die Analyse, wie sich lebende Zellen im Laufe der Zeit verändern, hilft Wissenschaftlern zu verstehen, wie sie sich in Gesundheit und Krankheit verhalten.
Mit Werkzeugen wie Phasenkontrast- oder hochauflösender Mikroskopie können Forscher diese Veränderungen verfolgen, ohne Farbstoffe oder Markierungen hinzuzufügen. Dies hält die Zellen in ihrem natürlichen Zustand und macht die Ergebnisse zuverlässiger.
Die Verfolgung von Zellen im Zeitverlauf hilft auch, Details zu erfassen, die sonst möglicherweise übersehen würden. Eine Zelle bewegt sich möglicherweise schneller als andere, teilt sich auf ungewöhnliche Weise oder reagiert stark auf einen Reiz. Die Aufzeichnung dieser subtilen Unterschiede vermittelt ein klareres Bild davon, wie sich Zellen unter realen Bedingungen verhalten.
Computer-Vision-Modelle wie Ultralytics YOLOv8 machen diesen Prozess schneller und konsistenter. Durch die Erkennung und Verfolgung einzelner Zellen über Bildsequenzen hinweg kann YOLOv8 Zellbewegungen, Teilungen und Interaktionen automatisch überwachen, selbst wenn sich Zellen überlappen oder ihre Form verändern.
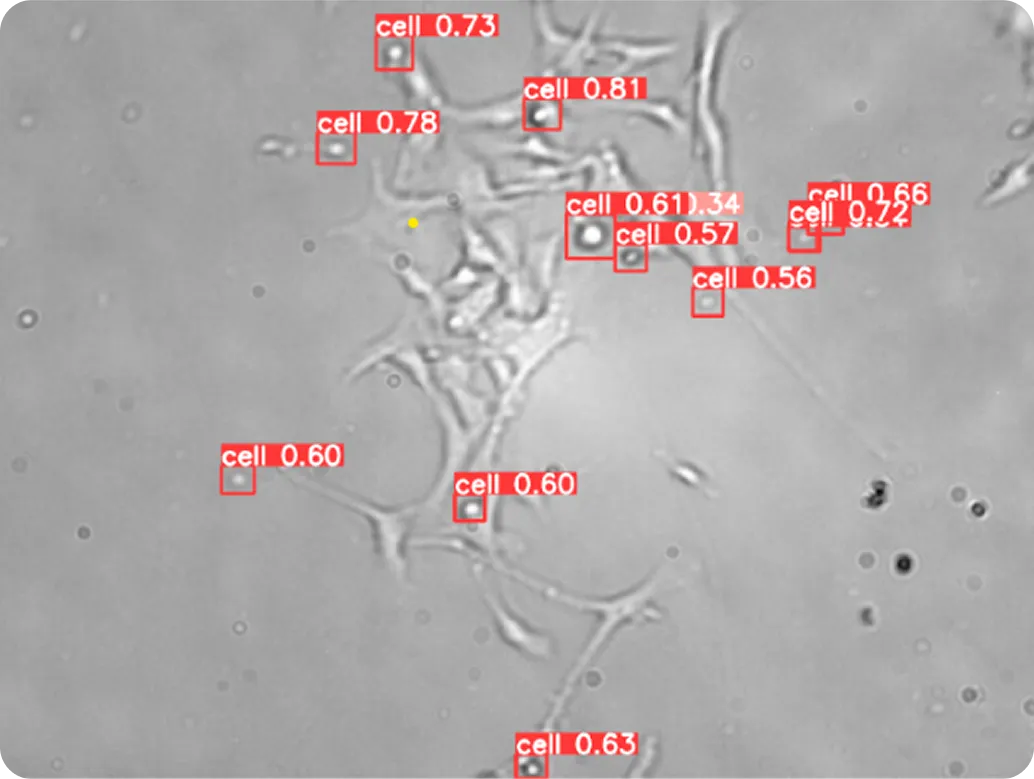
Erkenntnisse aus Computer-Vision-Modellen wie YOLO11 , die für die Segmentierung ganzer Zellen verwendet werden, können eine größere Wirkung entfalten, wenn sie mit Bioinformatik (dem Einsatz von Berechnungsmethoden zur Analyse biologischer Daten) und Multi-omics (der Integration von DNA-, RNA- und Proteininformationen) kombiniert werden. Zusammen bringen diese Methoden die Forschung über das Zeichnen von Zellgrenzen hinaus und zum Verständnis der Bedeutung dieser Grenzen.
Anstatt nur zu identifizieren, wo sich Zellen befinden, können Wissenschaftler untersuchen, wie sie interagieren, wie sich die Gewebestruktur bei Krankheiten verändert und wie kleine Veränderungen in der Zellform mit molekularer Aktivität zusammenhängen.
Nehmen wir eine Tumorprobe: Durch die Verknüpfung der Größe, Form oder Position von Krebszellen mit ihren molekularen Profilen können Forscher Korrelationen mit Genmutationen, Genexpression oder abnormaler Proteinaktivität finden. So werden aus statischen Bildern praktische Erkenntnisse, die helfen, die Genaktivität in Tumoren track , das Verhalten von Proteinen in Echtzeit abzubilden und Referenzatlanten zu erstellen, die Struktur und Funktion miteinander verbinden.
Hier sind einige der wichtigsten Vorteile der Verwendung von Computer Vision für die Zellsegmentierung:
Obwohl Computer Vision viele Vorteile für die Zellsegmentierung mit sich bringt, gibt es auch einige Einschränkungen. Hier sind einige Faktoren, die Sie beachten sollten:
Die nächste Generation der Zellsegmentierung wird wahrscheinlich durch Computer-Vision-Modelle definiert, die Geschwindigkeit, Genauigkeit und Skalierbarkeit kombinieren. Modelle wie U-Net waren sehr einflussreich, können aber rechenintensiv sein. Mit den Fortschritten in der Computer Vision bewegen sich die Forscher hin zu Modellen, die sowohl eine hohe Genauigkeit als auch eine Echtzeit-Performance liefern.
So können moderne Modelle wie Ultralytics YOLOv8 Mikroskopie-Bilder viel schneller segment als herkömmliche Ansätze und dennoch scharfe und genaue Grenzen erzeugen.
In einer kürzlich durchgeführten Studie zur Transmissionselektronenmikroskopie (TEM) zeigten die Leistungskennzahlen, dass YOLOv8 bis zu 43 Mal schneller als U-Net lief. Diese Art von Leistung ermöglicht die Analyse großer Datensätze in Echtzeit, was bei immer umfangreicheren Bildgebungsstudien immer wichtiger wird.

Diese Verbesserungen werden bereits in die Praxis umgesetzt. Plattformen wie Theiascope™ von Theia Scientific integrieren Ultralytics YOLO mit der Transmissionselektronenmikroskopie (TEM) und ermöglichen so eine konsistente und maßstabsgetreue Segmentierung von Nanostrukturen. Die Plattform nutzt Ultralytics YOLO für die Echtzeit-Erkennung und -Segmentierung und identifiziert automatisch Strukturen in TEM-Bildern, sobald diese aufgenommen werden, und wandelt sie in zuverlässige, analysierbare Daten um.
Die Zellsegmentierung spielt eine Schlüsselrolle in der modernen Mikroskopie und biomedizinischen Forschung. Sie ermöglicht es Wissenschaftlern, einzelne Zellen zu beobachten, track Fortschreiten von Krankheiten track und zu überwachen, wie sich Behandlungen auf das Zellverhalten auswirken. KI-Modelle wie YOLO11 machen diesen Prozess schneller und präziser. Durch die einfache Handhabung großer, komplexer Bilder stellen sie sicher, dass Experimente wiederholbar und skalierbar sind.
Treten Sie unserer Community bei und besuchen Sie unser GitHub-Repository, um mehr über KI zu erfahren. Auf unseren Lösungsseiten erfahren Sie mehr über Anwendungen wie KI in der Landwirtschaft und Computer Vision in der Logistik. Informieren Sie sich über unsere Lizenzoptionen und beginnen Sie noch heute mit der Entwicklung von Computer Vision!