Segmentación celular: Qué es y cómo la IA visual la mejora
Aprenda cómo funciona la segmentación celular y cómo la IA visual mejora el análisis de microscopía con aprendizaje profundo, métricas clave, conjuntos de datos y usos en el mundo real.
Aprenda cómo funciona la segmentación celular y cómo la IA visual mejora el análisis de microscopía con aprendizaje profundo, métricas clave, conjuntos de datos y usos en el mundo real.

Muchos avances en el descubrimiento de fármacos, la investigación del cáncer o la medicina personalizada comienzan con un reto clave: ver las células con claridad. Los científicos dependen de imágenes claras para track comportamiento celular, evaluar fármacos y explorar nuevas terapias.
Una sola imagen de microscopía puede contener miles de células superpuestas, lo que dificulta la visualización de los límites. La segmentación celular tiene como objetivo resolver esto separando claramente cada célula para un análisis preciso.
Pero la segmentación celular no siempre es sencilla. Un solo estudio puede producir miles de imágenes de microscopio detalladas, demasiadas para revisarlas a mano. A medida que los conjuntos de datos crecen, los científicos necesitan formas más rápidas y confiables de separar y estudiar las células.
De hecho, muchos científicos están adoptando la visión por ordenador, una rama de la IA que permite a las máquinas interpretar y analizar la información visual. Por ejemplo, modelos como Ultralytics YOLO11 que admiten la segmentación de instancias pueden entrenarse para separar células e incluso detect estructuras subcelulares. Esto permite realizar análisis precisos en cuestión de segundos, en lugar de horas, lo que ayuda a los investigadores a ampliar sus estudios de forma eficiente.
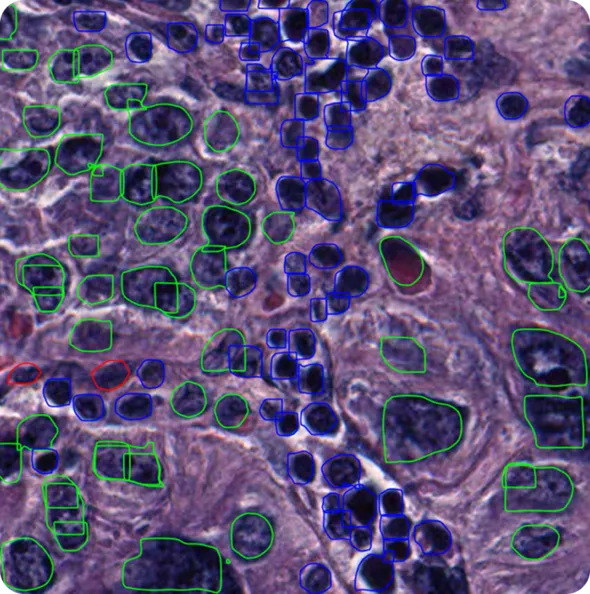
En este artículo, exploraremos cómo funciona la segmentación celular, cómo la visión artificial la mejora y dónde se aplica en el mundo real. ¡Empecemos!
Tradicionalmente, los científicos segmentaban las células a mano, trazándolas en imágenes de microscopía. Esto funcionaba bien para proyectos pequeños, pero era lento, inconsistente y propenso a errores. Con miles de células superpuestas en una sola imagen, el trazado manual se vuelve rápidamente abrumador y un importante cuello de botella.
La visión artificial proporciona una opción más rápida y fiable. Es una rama de la IA impulsada por el aprendizaje profundo, donde las máquinas aprenden patrones de grandes conjuntos de imágenes. En la investigación celular, esto significa que pueden reconocer y separar células individuales con gran precisión.
En concreto, los modelos de IA de visión como Ultralytics YOLO11 admiten tareas como la detección de objetos y la segmentación de instancias, y pueden entrenarse en conjuntos de datos personalizados para analizar células. La detección de objetos permite encontrar y etiquetar cada célula de una imagen, incluso cuando aparecen muchas juntas.
La segmentación de instancias va un paso más allá al dibujar límites precisos alrededor de cada célula, capturando sus formas exactas. La integración de estas capacidades de Visión Artificial en los flujos de trabajo de segmentación celular permite a los investigadores automatizar flujos de trabajo complejos y procesar imágenes de microscopía de alta resolución de forma eficiente.
Los métodos de segmentación celular han cambiado mucho a lo largo de los años. Las primeras técnicas de segmentación de imágenes funcionaban para imágenes simples, pero tenían dificultades a medida que los conjuntos de datos se hacían más grandes y las células se volvían más difíciles de distinguir.
Para superar estos límites, se desarrollaron enfoques más avanzados, lo que condujo a los modelos de visión artificial actuales que aportan velocidad, precisión y escalabilidad a los estudios de microbiología y microscopía.
A continuación, repasemos cómo han evolucionado los algoritmos de segmentación, desde los métodos básicos de umbralización hasta los modelos de deep learning de vanguardia y los pipelines híbridos.
Antes de los avances en la visión artificial, la segmentación celular se basaba en técnicas tradicionales de procesamiento de imágenes. Estos métodos se basaban en reglas y operaciones definidas manualmente, como la detección de bordes, la separación del primer plano del fondo o el suavizado de formas. A diferencia de los modelos de visión artificial, que pueden aprender patrones directamente de los datos, el procesamiento de imágenes depende de algoritmos fijos aplicados de la misma manera en todas las imágenes.
Uno de los primeros enfoques fue la umbralización, un método que separa las células del fondo comparando los niveles de brillo de los píxeles. Esto funciona bien cuando hay un fuerte contraste entre las células y su entorno.
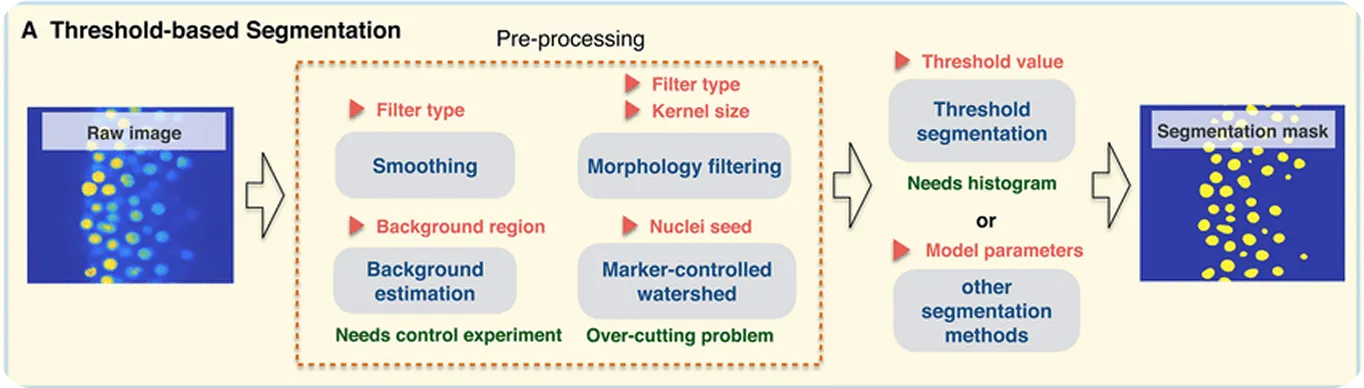
Para refinar los resultados, se utilizan operaciones morfológicas como la dilatación (expansión de formas) y la erosión (reducción de formas) para suavizar los bordes, eliminar el ruido o cerrar pequeños huecos. Para las células que se tocan o se superponen, una técnica llamada segmentación de watershed ayuda a separarlas trazando límites donde se encuentran las células.
Si bien estas técnicas tienen dificultades con casos complejos como células superpuestas o imágenes ruidosas, siguen siendo útiles para aplicaciones más sencillas y son una parte importante de la historia de la segmentación celular. Sin embargo, sus limitaciones impulsaron el campo hacia modelos basados en el deep learning, que ofrecen una precisión mucho mayor para las imágenes más complejas.
A medida que las técnicas de procesamiento de imágenes alcanzaron sus límites, la segmentación celular se orientó hacia enfoques basados en el aprendizaje. A diferencia de los métodos basados en reglas, los modelos de aprendizaje profundo identifican patrones directamente a partir de los datos, lo que los hace más adaptables a células superpuestas, formas variables y diferentes modalidades de imagen.
Las redes neuronales convolucionales (CNN) son una clase de arquitecturas de aprendizaje profundo muy utilizadas en visión por ordenador. Procesan las imágenes por capas: las primeras capas detect características sencillas como bordes y texturas, mientras que las capas más profundas capturan formas y estructuras más complejas. Este enfoque por capas hace que las CNN sean eficaces para muchas tareas visuales, desde el reconocimiento de objetos cotidianos hasta el análisis de imágenes biomédicas, pasando por el reconocimiento de patrones.
Modelos como YOLO11 se basan en estos principios de aprendizaje profundo. Amplían las arquitecturas basadas en CNN con técnicas de detección de objetos y segmentación de instancias en tiempo real, lo que permite localizar rápidamente las células y delinear sus límites.
Las canalizaciones híbridas mejoran la segmentación celular al combinar las fortalezas de múltiples métodos. Estos pueden incluir el procesamiento de imágenes clásico y los modelos de deep learning, o incluso diferentes modelos basados en deep learning que trabajan juntos.
Por ejemplo, un método puede mejorar o preprocesar las imágenes de microscopía para reducir el ruido y afinar los límites, mientras que otro modelo se utiliza para detect y segment las células. Al dividir las tareas de este modo, los enfoques híbridos mejoran la precisión, manejan imágenes complejas con mayor eficacia y hacen más fiables los estudios a gran escala.

Otro factor clave a considerar en la segmentación de células impulsada por la visión artificial son los datos de imagen. Los modelos de visión artificial dependen en gran medida de conjuntos de datos extensos y de alta calidad para aprender a identificar y separar las células con precisión.
Estos conjuntos de datos suelen consistir en imágenes de microscopía emparejadas con anotaciones. Tan importante como las imágenes sin procesar, la anotación de datos, o etiquetado, juega un papel crucial en la creación de conjuntos de datos eficaces, ya que le indica al modelo información clave.
Por ejemplo, si queremos entrenar un modelo como YOLO11 para segment células cancerosas a partir de imágenes de microscopía, necesitamos ejemplos etiquetados que muestren dónde empieza y acaba cada célula. Estas etiquetas sirven de guía y enseñan al modelo a reconocer la morfología y los límites de las células. Las anotaciones pueden dibujarse a mano o crearse con herramientas semiautomatizadas para ahorrar tiempo.
El tipo de anotación también depende de la tarea. Para la detección de objetos, se dibujan cuadros delimitadores alrededor de cada célula. Para la segmentación de instancias, las etiquetas son más como máscaras detalladas que trazan el contorno exacto de cada célula. Elegir el tipo correcto de anotación y datos de entrenamiento ayuda al modelo a aprender lo que necesita para el trabajo.
En general, la creación de conjuntos de datos de visión artificial puede ser difícil, especialmente cuando no existen colecciones de imágenes existentes o cuando el campo es muy singular y especializado. Pero en el campo de la investigación celular, existen dificultades técnicas que hacen que la recopilación y anotación de datos sean aún más complejas.
Las imágenes de microscopía pueden tener un aspecto muy diferente dependiendo del método de imagen celular. Por ejemplo, la microscopía de fluorescencia utiliza tintes que hacen que partes de una célula brillen. Estas imágenes de fluorescencia resaltan detalles que de otro modo serían difíciles de ver.
La anotación es otro desafío importante. Etiquetar miles de células a mano es lento y requiere experiencia en el dominio. Las células a menudo se superponen, cambian de forma o aparecen débiles, lo que facilita la aparición de errores. Las herramientas semiautomatizadas pueden acelerar el proceso, pero generalmente se necesita la supervisión humana para garantizar la calidad.
Para facilitar la carga de trabajo, los investigadores a veces utilizan anotaciones más simples, como marcadores de ubicación de interés que indican dónde están las células, en lugar de dibujar contornos completos. Aunque son menos precisos, estos marcadores siguen proporcionando una guía crucial para el entrenamiento.
Más allá de esto, el intercambio de datos en biología añade más complicaciones. Las preocupaciones sobre la privacidad, el consentimiento del paciente y las diferencias en los equipos de imagen entre los laboratorios pueden dificultar la creación de conjuntos de datos consistentes y de alta calidad.
A pesar de estos obstáculos, los conjuntos de datos de código abierto han supuesto una gran diferencia. Las colecciones públicas compartidas a través de plataformas como GitHub proporcionan miles de imágenes etiquetadas de muchos tipos de células y métodos de imagen, lo que ayuda a los modelos a generalizar mejor a los escenarios del mundo real.
Ahora que comprendemos mejor los datos y los métodos utilizados para segmentar células con visión artificial, veamos algunas de las aplicaciones reales de la segmentación celular y la visión artificial.
El análisis de células individuales, o el estudio de células individuales en lugar de muestras de tejido enteras, ayuda a los científicos a ver detalles que a menudo se pasan por alto a nivel general. Este enfoque se utiliza ampliamente en biología celular, descubrimiento de fármacos y diagnóstico para comprender cómo funcionan las células y cómo responden en diferentes condiciones.
Por ejemplo, en la investigación del cáncer, una muestra de tejido a menudo contiene una mezcla de células cancerosas, células inmunitarias y células de soporte (estromales). Observar sólo el tejido en su conjunto puede ocultar diferencias importantes, como la forma en que las células inmunitarias interactúan con los tumores o cómo se comportan las células cancerosas cerca de los vasos sanguíneos.
El análisis unicelular permite a los investigadores separar estos tipos celulares y estudiarlos individualmente, lo que resulta crucial para comprender las respuestas al tratamiento y la progresión de la enfermedad. Modelos como YOLO11, que admiten la segmentación de instancias, pueden detect cada célula y delinear su forma exacta, incluso en imágenes abarrotadas o superpuestas. Al convertir complejas imágenes de microscopía en datos estructurados, YOLO11 permite a los investigadores analizar miles de células de forma rápida y coherente.
Las células se dividen, se mueven y responden a su entorno de diferentes maneras. Analizar cómo cambian las células vivas a lo largo del tiempo ayuda a los científicos a comprender cómo se comportan en la salud y la enfermedad.
Con herramientas como el contraste de fase o la microscopía de alta resolución, los investigadores pueden seguir estos cambios sin añadir tintes ni etiquetas. Esto mantiene las células en su estado natural y hace que los resultados sean más fiables.
El seguimiento de las células a lo largo del tiempo también ayuda a capturar detalles que de otro modo podrían pasarse por alto. Una célula podría moverse más rápido que otras, dividirse de una manera inusual o responder fuertemente a un estímulo. El registro de estas sutiles diferencias proporciona una imagen más clara de cómo se comportan las células en condiciones reales.
Modelos de visión por ordenador como Ultralytics YOLOv8 hacen que este proceso sea más rápido y coherente. Mediante la detección y el seguimiento de células individuales a través de secuencias de imágenes, YOLOv8 puede supervisar los movimientos, las divisiones y las interacciones celulares de forma automática, incluso cuando las células se superponen o cambian de forma.
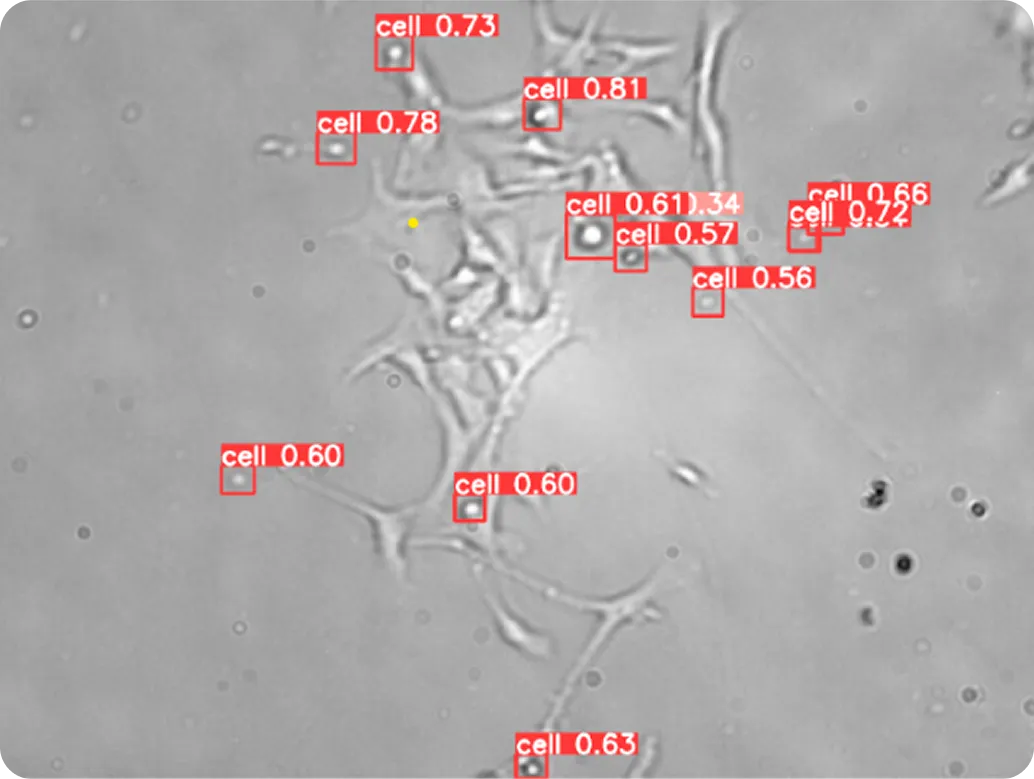
Los modelos de visión computerizada como YOLO11 , utilizados para la segmentación de células completas, pueden tener una mayor repercusión si se combinan con la bioinformática (el uso de métodos computacionales para analizar datos biológicos) y la multiómica (la integración de información sobre ADN, ARN y proteínas). Juntos, estos métodos hacen que la investigación vaya más allá del trazado de los límites celulares y se adentre en la comprensión del significado de esos límites.
En lugar de solo identificar dónde están las células, los científicos pueden estudiar cómo interactúan, cómo cambia la estructura de los tejidos en la enfermedad y cómo los pequeños cambios en la forma de las células se conectan con la actividad molecular.
Consideremos una muestra de tumor: al vincular el tamaño, la forma o la posición de las células cancerosas con sus perfiles moleculares, los investigadores pueden encontrar correlaciones con mutaciones genéticas, expresión génica o actividad proteica anormal. De este modo, las imágenes estáticas se convierten en conocimientos prácticos que ayudan a track la actividad de los genes en los tumores, cartografiar el comportamiento de las proteínas en tiempo real y crear atlas de referencia que relacionan la estructura con la función.
Estos son algunos de los principales beneficios de utilizar la visión artificial para la segmentación celular:
Si bien la visión artificial aporta muchos beneficios a la segmentación celular, también tiene algunas limitaciones. Aquí hay algunos factores a tener en cuenta:
La próxima generación de segmentación celular probablemente estará definida por modelos de visión artificial que combinen velocidad, precisión y escalabilidad. Modelos como U-Net han sido muy influyentes, pero pueden ser computacionalmente exigentes. Con los avances en la visión artificial, los investigadores están avanzando hacia modelos que ofrecen tanto alta precisión como rendimiento en tiempo real.
Por ejemplo, los modelos más avanzados, como Ultralytics YOLOv8 , pueden segment imágenes de microscopía mucho más rápido que los métodos tradicionales, sin dejar de producir límites nítidos y precisos.
En un reciente estudio de microscopía electrónica de transmisión (TEM), las métricas de rendimiento mostraron que YOLOv8 funcionaba hasta 43 veces más rápido que U-Net. Este tipo de rendimiento permite analizar grandes conjuntos de datos en tiempo real, algo cada vez más importante a medida que aumenta el tamaño de los estudios de imagen.

Estas mejoras ya se están poniendo en práctica. Plataformas como Theiascope™ de Theia Scientific integran los modelosYOLO Ultralytics con la microscopía electrónica de transmisión (TEM), lo que permite segmentar estructuras a nanoescala de forma coherente y a escala. La plataforma utiliza modelos Ultralytics YOLO para la detección y segmentación en tiempo real, identificando automáticamente estructuras en imágenes TEM a medida que se capturan y convirtiéndolas en datos fiables y listos para analizar.
La segmentación celular desempeña un papel fundamental en la microscopía moderna y la investigación biomédica. Permite a los científicos observar células individuales, track progresión de enfermedades y controlar cómo afectan los tratamientos al comportamiento celular. Los modelos de IA de visión como YOLO11 hacen que este proceso sea más rápido y preciso. Al manejar con facilidad imágenes grandes y complejas, garantizan la repetibilidad y escalabilidad de los experimentos.
Únase a nuestra comunidad y visite nuestro repositorio de GitHub para obtener más información sobre la IA. Explore nuestras páginas de soluciones para obtener más información sobre aplicaciones como la IA en la agricultura y la visión artificial en la logística. Consulte nuestras opciones de licencia y comience a construir con visión artificial hoy mismo.