細胞セグメンテーションの仕組み、およびVision AIが深層学習、主要指標、データセット、および実際の使用法で顕微鏡分析をどのように改善するかについて学びましょう。
細胞セグメンテーションの仕組み、およびVision AIが深層学習、主要指標、データセット、および実際の使用法で顕微鏡分析をどのように改善するかについて学びましょう。

創薬、がん研究、個別化医療における画期的な進歩の多くは、細胞を鮮明に見るという一つの重要な課題から始まる。科学者は、細胞の挙動をtrack し、薬剤を評価し、新しい治療法を探求するために、鮮明な画像に依存している。
単一の顕微鏡画像には、数千もの重複する細胞が含まれている可能性があり、境界が見えにくくなっています。細胞セグメンテーションは、各細胞を明確に分離して正確な分析を行うことで、この問題を解決することを目的としています。
しかし、細胞のセグメンテーションは必ずしも単純ではありません。1回の研究で、数千枚の詳細な顕微鏡画像が生成されることがあり、手作業で確認するには多すぎます。データセットが拡大するにつれて、科学者たちは細胞を分離して研究するための、より高速で信頼性の高い方法を必要としています。
実際、多くの科学者が、機械が視覚情報を解釈・分析できるようにするAIの一分野であるコンピューター・ビジョンを採用している。例えば Ultralytics YOLO11のようなインスタンスのセグメンテーションをサポートするモデルは、細胞を分離し、細胞内構造をdetect ようにトレーニングすることができる。これにより、数時間ではなく数秒で正確な分析が可能になり、研究者が効率的に研究をスケールアップできるようになる。
この記事では、細胞セグメンテーションの仕組み、コンピュータビジョンによる改善、そして現実世界での応用について解説します。それでは始めましょう!
従来、科学者は顕微鏡画像で細胞を手動でトレースすることにより、細胞をセグメント化していました。これは小規模なプロジェクトには適していましたが、時間がかかり、一貫性がなく、エラーが発生しやすいものでした。1枚の画像に何千もの重複する細胞がある場合、手動トレースはすぐに手に負えなくなり、大きなボトルネックになります。
コンピュータビジョンは、より高速で信頼性の高いオプションを提供します。これは、深層学習を利用したAIの一分野であり、機械は大量の画像セットからパターンを学習します。セル研究では、これは、個々のセルを高精度で認識および分離できることを意味します。
具体的には、Ultralytics YOLO11 ようなビジョンAIモデルは、オブジェクト検出やインスタンス分割などのタスクをサポートし、細胞を分析するためのカスタムデータセットで学習させることができる。物体検出は、多数の細胞が一緒に写っている場合でも、画像内の各細胞を見つけてラベル付けすることを可能にする。
インスタンスセグメンテーションは、各細胞の正確な形状を捉え、それぞれの周囲に正確な境界線を描くことで、さらに一歩進んでいます。これらのVision AI機能を細胞セグメンテーションパイプラインに統合することで、研究者は複雑なワークフローを自動化し、高解像度の顕微鏡画像を効率的に処理できます。
細胞セグメンテーションの方法は、長年にわたって大きく変化してきました。初期の画像セグメンテーション技術は、単純な画像では機能しましたが、データセットが大きくなり、細胞の区別が難しくなるにつれて苦戦しました。
これらの制限を克服するために、より高度なアプローチが開発され、今日のコンピュータビジョンモデルにつながり、微生物学および顕微鏡研究に速度、精度、スケーラビリティをもたらしています。
次に、基本的な閾値処理法から最先端の深層学習モデルやハイブリッドパイプラインまで、セグメンテーションアルゴリズムがどのように進化してきたかを順を追って説明します。
コンピュータビジョンの進歩以前は、細胞セグメンテーションは従来からの画像処理技術に依存していました。これらの手法は、エッジの検出、前景と背景の分離、または形状の平滑化など、手動で定義されたルールと操作に依存していました。データから直接パターンを学習できるコンピュータビジョンモデルとは異なり、画像処理はすべての画像に同じ方法で適用される固定アルゴリズムに依存します。
最も初期のアプローチの1つは、ピクセルの明るさのレベルを比較してセルを背景から分離する閾値処理でした。これは、セルとその周囲の間に強いコントラストがある場合に有効です。
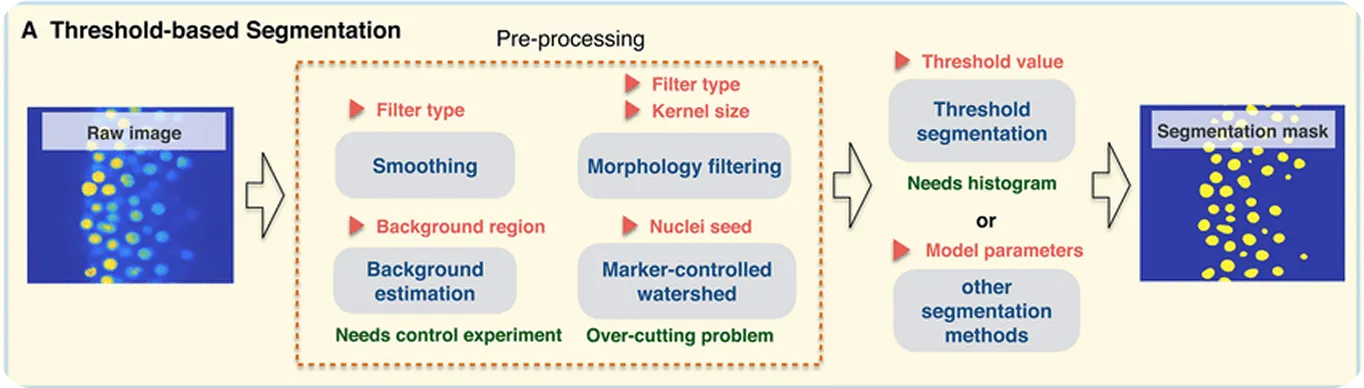
結果を改善するために、膨張(形状の拡大)や収縮(形状の縮小)などのモルフォロジー演算を使用して、エッジを滑らかにしたり、ノイズを除去したり、小さなギャップを閉じたりします。接触または重複するセルについては、分水嶺セグメンテーションと呼ばれる手法を使用して、セルが接する境界線を描画することにより、セルを分離します。
これらの手法は、細胞の重複やノイズの多い画像のような複雑なケースには対応できませんが、より単純なアプリケーションには依然として有用であり、細胞セグメンテーションの歴史において重要な部分を占めています。しかし、その限界から、この分野は深層学習ベースのモデルへと移行し、より困難な画像に対してはるかに高い精度を実現しています。
画像処理技術が限界に達すると、細胞のセグメンテーションは学習ベースのアプローチに移行しました。ルールベースの方法とは異なり、深層学習モデルはデータから直接パターンを識別するため、重複する細胞、可変形状、および異なるイメージングモダリティへの適応性が高まります。
畳み込みニューラルネットワーク(CNN)は、コンピュータビジョンで広く使われているディープラーニングアーキテクチャの一種である。初期の層はエッジやテクスチャのような単純な特徴をdetect し、より深い層はより複雑な形状や構造を捉える。このレイヤーアプローチにより、CNNは、パターン認識による日常的な物体の認識から生物医学的画像の分析まで、多くの視覚タスクに有効である。
YOLO11 ようなモデルは、このようなディープラーニングの原理に基づいて構築されている。CNNベースのアーキテクチャーを、リアルタイムの物体検出とインスタンスのセグメンテーションのための技術で拡張し、細胞の位置を素早く特定し、その境界の輪郭を描くことを可能にしている。
ハイブリッドパイプラインは、複数の手法の強みを組み合わせることで、細胞セグメンテーションを向上させます。これには、従来の画像処理や深層学習モデル、さらには連携して動作するさまざまな深層学習ベースのモデルが含まれる場合があります。
例えば、ある手法では顕微鏡画像を強調したり前処理してノイズを減らし境界を鮮明にし、別のモデルでは細胞のdetect segment 行う。このようにタスクを分割することで、ハイブリッド・アプローチは精度を向上させ、複雑な画像をより効果的に扱い、大規模研究をより信頼性の高いものにする。

コンピュータビジョン駆動の細胞セグメンテーションで考慮すべきもう1つの重要な要素は、画像データです。コンピュータビジョンモデルは、細胞を正確に識別および分離する方法を学習するために、大規模で高品質のデータセットに大きく依存しています。
これらのデータセットは通常、顕微鏡画像とアノテーションで構成されています。生の画像と同様に重要なデータアノテーション、つまりラベル付けは、モデルに重要な情報を伝えるため、効果的なデータセットの作成において重要な役割を果たします。
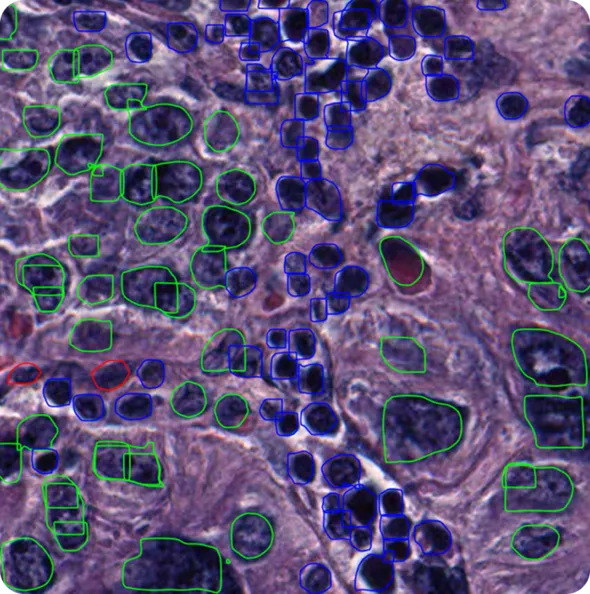
例えば、YOLO11 ようなモデルを学習させ、顕微鏡画像からがん細胞をsegment せたい場合、各細胞がどこで始まり、どこで終わるかを示すラベル付きサンプルが必要になる。これらのラベルはガイドとして機能し、モデルに細胞の形態と境界を認識する方法を教える。注釈は手作業で描くことも、時間を節約するために半自動化ツールで作成することもできる。
アノテーションの種類もタスクによって異なります。物体検出の場合、各セルを囲むバウンディングボックスが描画されます。インスタンスセグメンテーションの場合、ラベルは各セルの正確な輪郭をトレースする詳細なマスクのようなものです。適切な種類のアノテーションとトレーニングデータを選択することで、モデルはジョブに必要なものを学習できます。
一般に、コンピュータビジョンデータセットの構築は、特に既存の画像コレクションがない場合や、分野が非常にユニークで特殊な場合には困難になる可能性があります。しかし、細胞研究の分野では、データ収集とアノテーションをさらに複雑にする技術的な困難があります。
顕微鏡画像は、細胞イメージングの方法によって大きく異なる場合があります。たとえば、蛍光顕微鏡法では、細胞の一部を発光させる色素を使用します。これらの蛍光画像は、通常は見えにくい詳細を強調表示します。
アノテーションもまた、大きな課題です。何千もの細胞を手作業でラベル付けするのは時間がかかり、専門知識が必要です。細胞はしばしば重なり合ったり、形を変えたり、かすかに見えたりするため、間違いが起こりやすくなります。半自動化ツールはプロセスをスピードアップできますが、品質を確保するには通常、人間の監視が必要です。
作業負荷を軽減するために、研究者は完全なアウトラインを描画するのではなく、細胞の位置を示す関心領域マーカーなどのより単純なアノテーションを使用することがあります。精度は劣りますが、これらのマーカーはトレーニングに不可欠なガイダンスを提供します。
さらに、生物学におけるデータ共有は、問題を複雑にします。プライバシーに関する懸念、患者の同意、研究室間の画像処理装置の違いにより、一貫性のある高品質なデータセットの構築が困難になる可能性があります。
これらの障害にもかかわらず、オープンソースデータセットは大きな変化をもたらしました。GitHubのようなプラットフォームを通じて共有される公開コレクションは、多くの細胞タイプとイメージング方法にわたる数千ものラベル付き画像を提供し、モデルが実際のシナリオにより良く一般化するのに役立ちます。
コンピュータビジョンによる細胞のセグメンテーションに使用されるデータと手法について理解が深まったところで、細胞のセグメンテーションとコンピュータビジョンの実際の応用例をいくつか見ていきましょう。
シングルセル解析、つまり組織サンプル全体ではなく個々の細胞を研究することで、科学者はより広いレベルでは見逃されがちな詳細を把握できます。このアプローチは、細胞がさまざまな条件下でどのように機能し、応答するかを理解するために、細胞生物学、創薬、診断で広く使用されています。
例えば、がん研究では、組織サンプルには、がん細胞、免疫細胞、および支持細胞(間質細胞)が混在していることがよくあります。組織全体だけを見ると、免疫細胞が腫瘍とどのように相互作用するか、または血管近くのがん細胞がどのように振る舞うかなど、重要な違いが隠れてしまう可能性があります。
単一細胞解析により、研究者はこれらの細胞タイプを分離して個別に研究することができ、これは治療反応や疾患の進行を理解する上で極めて重要である。インスタンスのセグメンテーションをサポートするYOLO11ようなモデルは、混雑した画像や重なり合った画像であっても、各細胞をdetect し、その正確な形状の輪郭を描くことができる。複雑な顕微鏡画像を構造化されたデータに変換することで、YOLO11 11は研究者が何千もの細胞を迅速かつ一貫して分析することを可能にします。
細胞は、さまざまな方法で分裂、移動し、周囲の環境に反応します。生きた細胞が時間の経過とともにどのように変化するかを分析することで、科学者は健康と病気における細胞の挙動を理解できます。
位相差顕微鏡や高解像度顕微鏡のようなツールを使用することで、研究者は色素やラベルを追加せずにこれらの変化を追跡できます。これにより、細胞は自然な状態に保たれ、結果の信頼性が高まります。
細胞を経時的に追跡することで、見逃される可能性のある詳細を捉えることもできます。ある細胞は他の細胞よりも速く移動したり、通常とは異なる方法で分裂したり、刺激に強く反応したりする場合があります。これらのわずかな違いを記録することで、実際の条件下での細胞の挙動をより明確に把握できます。
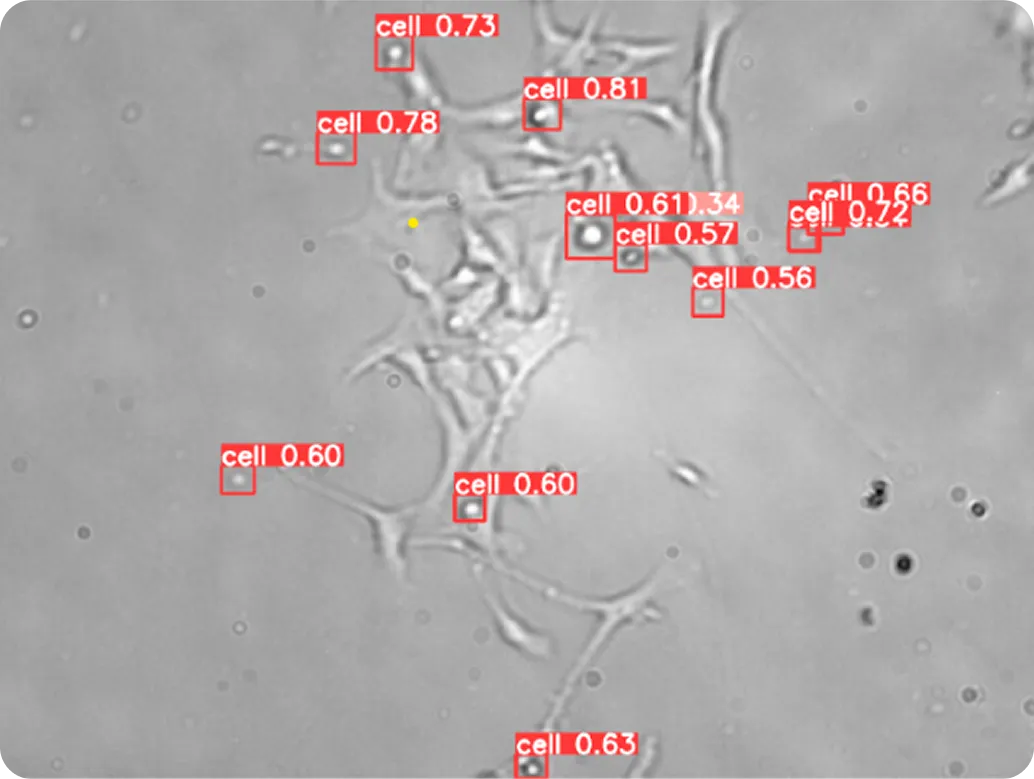
以下のようなコンピュータビジョンモデル Ultralytics YOLOv8は、このプロセスをより迅速かつ一貫性のあるものにします。YOLOv8 、画像シーケンス全体にわたって個々の細胞を検出し追跡することにより、細胞が重なり合ったり形状が変化した場合でも、細胞の動き、分裂、相互作用を自動的にモニターすることができる。
全細胞のセグメンテーションに使われるYOLO11 ようなコンピューター・ビジョン・モデルからの洞察は、バイオインフォマティクス(生物学的データを解析するための計算手法)やマルチオミクス(DNA、RNA、タンパク質情報の統合)と組み合わせることで、より大きなインパクトを与えることができる。これらの方法を組み合わせることで、研究は細胞の境界線を描くだけでなく、その境界線が何を意味するのかを理解することにつながる。
科学者は、細胞がどこにあるかを特定するだけでなく、細胞がどのように相互作用するか、病気で組織構造がどのように変化するか、および細胞形状の小さな変化が分子活動にどのように関連するかを研究できます。
腫瘍サンプルを考えてみよう。がん細胞の大きさ、形、位置を分子プロファイルと結びつけることで、研究者は遺伝子変異、遺伝子発現、異常なタンパク質活性との相関を見つけることができる。これは、静的な画像を実用的な洞察に変え、腫瘍全体の遺伝子活性をtrack し、タンパク質の挙動をリアルタイムでマッピングし、構造と機能を結びつける参照アトラスを構築するのに役立つ。
細胞セグメンテーションにコンピュータビジョンを使用する主な利点を以下に示します。
コンピュータビジョンは細胞のセグメンテーションに多くの利点をもたらしますが、いくつかの制限もあります。留意すべき点がいくつかあります。
次世代の細胞セグメンテーションは、速度、精度、およびスケーラビリティを兼ね備えたコンピュータビジョンモデルによって定義される可能性があります。U-Netなどのモデルは非常に影響力がありましたが、計算負荷が高くなる可能性があります。コンピュータビジョンの進歩により、研究者は高い精度とリアルタイムパフォーマンスの両方を提供するモデルに移行しています。
例えば、Ultralytics YOLOv8 ような最先端のモデルは、シャープで正確な境界線を生成しながらも、従来のアプローチよりもはるかに高速に顕微鏡画像をsegment することができる。
最近の透過型電子顕微鏡(TEM)研究では、パフォーマンス・メトリクスの結果、YOLOv8 U-Netより最大43倍高速に動作した。このような性能により、大規模なデータセットをリアルタイムで解析することが可能になり、イメージング研究が大規模化するにつれて、その重要性が増している。

これらの改善は、すでに実用化されている。Theia Scientific社のTheiascope™のようなプラットフォームは、Ultralytics YOLO モデルと透過型電子顕微鏡(TEM)を統合し、ナノスケールの構造を一貫したスケールでセグメンテーションできるようにします。このプラットフォームは、リアルタイムの検出とセグメンテーションにUltralytics YOLO モデルを使用し、撮影されたTEM画像内の構造を自動的に識別し、信頼性の高い、すぐに分析可能なデータに変換します。
細胞のセグメンテーションは、現代の顕微鏡と生物医学研究において重要な役割を果たしている。これにより、科学者は個々の細胞を観察し、病気の進行をtrack し、治療が細胞の挙動にどのように影響するかをモニターすることができる。YOLO11 ようなビジョンAIモデルは、このプロセスをより迅速かつ正確に行う。大きく複雑な画像を簡単に扱うことで、実験の再現性と拡張性を保証します。
コミュニティに参加し、GitHubリポジトリにアクセスして、AIについてさらに学びましょう。ソリューションページでは、農業におけるAIや物流におけるコンピュータビジョンなどのアプリケーションについて詳しくご紹介しています。ライセンスオプションを確認して、今日からコンピュータビジョンの構築を始めましょう。